DNAmark
– en DNA referencedatabase for danske arter
Baggrund
Indtil for ganske nylig blev overvågning af biodiversitet hovedsagelig udført ved direkte observationer samt ved indsamling og morfologisk identificering af organismer. Men nye teknikker er ved at vinde indpas, og især eDNA (environmental DNA), på dansk miljø-DNA, har de sidste år revolutioneret kortlægningen af biodiversitet. Forskere over hele verden har vist, at miljø-DNA er en sensitiv, økonomisk og effektiv metode til at afdække arters tilstedeværelse via for eksempel prøver af sediment, jord, ferskvand, havvand, fæces, samt fra multi-individprøver af invertebrater fanget i net eller faldfælder og af maveindhold fra blodsugende og kødædende invertebrater. I sådanne studier er miljø-DNA blandt andet blevet brugt til at afsløre tilstedeværelse af sjældne arter og indikatorarter, bestemme biodiversitet både indenfor og imellem lokaliteter, til tidlig opdagelse af invasive arter samt til at vurdere effekter af klimaforandringer.
En konsekvens af den nuværende måde at kortlægge biodiversitet på er, at prioritering af arealer og evaluering af virkemidler i naturbeskyttelsen primært er baseret på floraen og fuglefaunaen. Dette skyldes, at langt de fleste organismegrupper er ineffektive som indikatorer, fordi de er vanskelige at identificere for ikke-specialister, eller fordi arterne er for begrænsede og uforudsigelige i deres forekomst. Det er navnlig et problem for ekstraordinært diverse grupper som insekter og svampe. Problemet med den tilgang er, at ingen artsgrupper, heller ikke fugle eller planter, fungerer som indikatorartsgruppe for den samlede biodiversitet af alle organismer. Det betyder, at naturbeskyttelsens prioritering af arealer ofte rammer skævt i forhold til sikring af uerstattelige bestande af arter, som er vanskelige at erkende, især arter af meget artsrige grupper som insekter og svampe. Endvidere vil konkrete tiltag i naturforvaltningen, som for eksempel regulering af vandstand eller græsningstryk, ofte have positiv virkning på én organismegruppe, men negativ på andre grupper. Beslutninger om prioritering og indsatser træffes altså på et mangelfuldt grundlag. En oplagt løsning er at foretage en samtidig kortlægning af biologisk mangfoldighed af alle – eller i hvert fald mange og forskelligartede – taxonomiske grupper, kaldet multi-taxon biodiversitet. Det er først med udviklingen af miljø-DNA-analyser, at dette er blevet muligt. Derfor har metoden potentiale til at gøre naturbeskyttelse og -forvaltning målrettet og effektiv i forhold til den samlede biodiversitet og ikke blot en begrænset del af den.
På trods af, at miljø-DNA har potentiale til at blive et nyt effektivt redskab til kortlægning af biodiversitet, så lader implementering af miljø-DNA i naturbeskyttelse vente på sig. Vi er begyndt at se et skift i forskningen fra at have haft fokus på metodeudvikling og -evaluering, til et begyndende fokus på anvendelse i naturovervågning og -forvaltning, men en egentlig implementering af miljø-DNA er endnu ikke sket noget sted i verden. Implementering kræver først og fremmest, at miljø-DNA integreres med konventionelle metoder, både for at bygge på eksisterende viden, men også for at afdække metodens stærke og svage sider og dermed validere miljø-DNA som et pålideligt og omkostningseffektivt redskab til den givne opgave. Endvidere kræver implementering et standardiseret set-up, således at resultater bliver sammenlignelige både indenfor og på tværs af projekter. Men først og fremmest kræves en veludviklet DNA-referencedatabase. Uden en referencedatabase kan man nemlig ikke ‘oversætte’ de fundne miljø-DNA-sekvenser til kendte arter. Dernæst kræver implementering af miljø-DNA viden om, hvilke miljøprøvetyper der skal tages, og hvordan de skal indsamles for bedst muligt at repræsentere den samlede biodiversitet i et givent økosystem. Dette kan kun opnås, hvis vi har viden om, hvordan biodiversiteten fordeler sig i forhold til økologisk rum, herunder levesteder, som er fordelt meget ujævnt i tid og rum. Endelig medfører multi-taxon biodiversitet nye udfordringer ved at inkludere dårligt kendte organismegrupper, hvor arternes generelle hyppighed er ukendt, og dermed deres unikke bidrag til biodiversiteten regionalt eller globalt. Ydermere, er det af afgørende betydning for multi-taxon biodiversiteten at fordelingen indenfor og mellem artsgrupper estimeres. Der er derfor brug for udvikling af metoder til at håndtere disse udfordringer.
Projektet beskrivelse
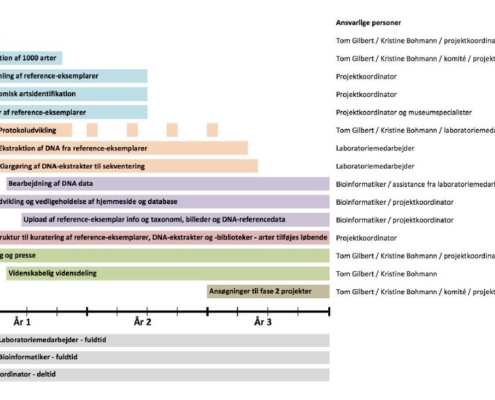
DNAmark vil i løbet af en 3-årig projektperiode skabe solidt grundlag for arbejdet med molekylært biodiversitets- og overvågningsarbejde i Danmark. Projektet har opstart 1. maj 2017 og består af to overordnede arbejdspakker, Arbejdspakke 1 og Arbejdspakke 2, der omhandler to forskellige, men direkte relaterede aspekter, der er nødvendige for fremtidig brug af miljø-DNA i dansk naturforvaltning: (i) Opbygning af en dansk DNA-referencedatabase, og (ii) udvikling af miljø-DNA felt-, laboratorie- og analysemetodik til at kortlægge multi-taxon biodiversitet (inklusiv artsrige, men vanskelige grupper som spindlere, insekter og svampe) i terrestriske økosystemer langs hele gradienten i successionstrin fra klitlavning over højmose til gamle krat og urskov.
Fordi kvaliteten af DNA-referencedatabasen er altafgørende for miljø-DNA-metoders styrke, etablerer vi i Arbejdspakke 1 en omfattende, standardiseret og fremtidssikret DNA-referencedatabase (kaldet DNAmark ligesom projektet). Desuden genererer vi protokoller og referencedata for mindst 1000 danske arter på tværs af livets træ (dyr, planter, alger og svampe) udvalgt i samarbejde med en komité af interesserede parter, herunder taxonomiske eksperter. Ved projektets afslutning har vi etableret en solid dansk DNA-referencedatabase på tværs af danske arter, samt protokoller og arbejdsgange for generering af referencedata for hver taxonomiske gruppe. Dermed skaber vi et solidt grundlag for fremtidig udbygning af DNAmark-referencedatabasen.
I Arbejdspakke 2 udvikler vi en robust indsamlingsmetodik til fremtidig anvendelse af miljø-DNA i dansk naturovervågning og -forvaltning. Vi udvikler metoder til omkostningseffektiv molekylær kortlægning af invertebrat-, plante- og svampe-diversitet i prøver af jord, levende og døde plantedele m.v. Formålet er at opnå et repræsentativt estimat på den samlede biodiversitet af alle organismegrupper, navnlig de mest artsrige, nemlig svampene og insekterne. Konkret består arbejdspakken af to dele: I Arbejdspakke 2A udarbejder vi en protokol til molekylær og analytisk opgørelse af terrestrisk multi-taxon biodiversitet. Her kortlægger vi (i) det minimale antal jordprøver i tid og rum, der er tilstrækkeligt til at sige noget afgørende om en lokalitets samlede diversitet af invertebrater, planter og svampe, (ii) antallet af nødvendige laboratorie-replikater, (iii) udvikler analytiske metoder til estimering af artsfordelingen indenfor og mellem taxonomiske grupper, og (iv) udvikler en prøvetagningsprotokol, der dækker variation i biodiversitet knyttet til kryptiske levesteder og usædvanlige kulstofkilder, som tiltager i betydning med økosystemets modenhed og kompleksitet, og som ikke kan detekteres fra jordprøver alene. I Arbejdspakke 2B omsætter vi multi-taxon biodiversitet til naturbevaringssværdi i form af “eneståenhed”. Det gør vi ved at sætte den lokale biodiversitet i relation til biodiversiteten på regional eller national skala og ved at vurdere det unikke bidrag fra hver lokalitet til helheden.
Med de to arbejdspakker i DNAmark faciliterer vi den fremtidige brug af miljø-DNA i dansk naturforvaltning, sætter en høj international standard og skaber basis for, at miljø-DNA kan bidrage til dansk naturforvaltning som et økonomisk, stærkt og pålideligt værktøj. Den 3-årige projektperiode danner grundlag for fremtidige projekter omhandlende udbygning af DNA-referencedatabasen og for projekter, der bruger DNA-referencedatabasen og de udviklede metoder i anvendte miljø-DNA-projekter i Danmark. Ideelt set udgør den 3-årige projektperiode dermed fase 1 i et flerfase-projekt, som fremtidige anvendte miljø-DNA-projekter kan bygges på. En naturlig måde at udbygge projektet på vil være at relevante danske forskningsgrupper ansøger om fase 2-projekter i projektets tredje år.
Status 2019
Denne årsrapport gør rede for udfordringer, resultater og fremskridt i DNAmark-projektets første 2,5 år, og giver bud på fremtidige perspektiver og projekter.
Arbejdspakke 1 har de sidste 2,5 år udviklet arbejdsgange og genereret referencedata for så forskellige prøvetyper som muligt for danske dyr, planter og svampe på tværs af taksonomiske grupper og af særlig interesse for dansk naturovervågning. Arterne er udvalgt og indsamlet med hjælp fra en komité af interesserede parter, som består af blandt andet konsulenter, kuratorer, miljøDNA-forskere og naturforvaltere. Vi har i skrivende stund indsamlet prøver fra over 1300 forskellige danske arter. Disse er fordelt på 188 forskellige taksonomiske ordner og spænder over ferskvandsindikatorarter, tørvemosearter, sjældne orkideer, samtlige danske arter af padder og krybdyr, næsten alle pattedyrsarter og halvdelen af danske fiskearter. Vi har ekstraheret DNA fra over 1000 arter, og nye protokoller og arbejdsgange er udviklet, således at vi kan generere DNA-referencedata fra forskellige taksonomiske grupper og prøvetyper. Over halvdelen af de 1000 arter er DNA-sekventeret og deres mitokondrielle genomer samlet.
De resterende arter er i kø til sekventering eller befinder sig på forskellige stadier i den bioinformatiske pipeline. Vi er ved at lægge sidste hånd på udviklingen af den hjemmeside, hvorfra de danske data kan tilgås. Hjemmesiden vil skabe overblik og let adgang til det genererede DNAmark-referencedata i NCBI Genbank. Mange projekter har allerede vist interesse for det genererede referencedata, for eksempel Einar Eg Nielsens og Philip Francis Thomsens marine miljøDNA-projekter. Vi håber at kunne fortsætte arbejdet med DNAmark DNA-referencedatabasen efter projektets udløb.
Arbejdspakke 2 har videreudviklet miljøDNA-teknikker til et praktisk værktøj til objektivt at opgøre biodiversitet. Målet er, at man i fremtiden kan anvende dette værktøj i prioritering af arealer og i evaluering af virkemidler i naturbeskyttelse. I projektets første år fokuserede vi på 10 udvalgte prøveflader ud af Biowide-projektets 130 prøveflader, som repræsenterede forskellige økosystemer fra simple til komplekse samt forskellige abiotiske miljøer. I disse 10 udvalgte prøveflader, har vi indsamlet prøver fra mere end 300 forskellige substrater (levesteder), som vi har grund til at tro er levested for forskellige puljer af arter.
Dertil er der i 5 af de 10 prøveflader indsamlet jordprøver i variable mængder på forskellige årstider og ekstraheret med forskellige ekstraktionskits for at klarlægge, hvilke variable der er mest betydende for variationen i den registrerede biodiversitet. Dette med henblik på at optimere og effektivisere prøvetagning og reducere omkostninger. I det forgangne år har vi analyseret resultaterne af DNAekstraktion og –sekvensering fra de mange prøver. Vi tør godt løfte sløret for at miljøDNA ekstraheret fra jord alene er utilstrækkeligt til at estimere den samlede biodiversitet i økosystemer, bortset fra på agerjord.
Der er unikke bidrag til den samlede biodiversitet fra alle substratprøver. Den gode nyhed er, at prøver af et enkelt substrat, nemlig bladførne fra jordoverfladen, ser ud til både at være jordprøver overlegne og at opfange en meget stor del af den samlede biodiversitet. I skovøkosystemer skal denne prøvetype dog suppleres med prøver af døde træstammer. Disse foreløbige resultater lover godt for udviklingen af en praktisk indsamlingsprotokol for omkostningseffektiv indsamling af DNA-prøver i biodiversitetsspørgsmål.
Selvom analyserne ikke er kørt til ende, kan vi afsløre at det måske vil være muligt at opnå repræsentative biodiversitetsprøver (jord) med mindre prøvevolumener end anvendt i Biowide-projektet. Vores mål er, at DNAmark-projektet skal være synligt for og anvendes af omverdenen, både af nationale og internationale fagfolk og dele af offentligheden, som kunne have interesse i og gavn af projektet. Vi formidler derfor projektet bredt både videnskabeligt og populærvidenskabeligt.
Status 2018
DNAmark-projektet etablerer med støtte fra AAGE V. JENSEN NATURFOND en national DNA-referencedatabase. I projektets første tre år bliver der lavet DNA-referencedata for 1000 danske dyr, planter og svampe. DNAmark skaber dermed grundlag for molekylært biodiversitets- og naturovervågningsarbejde i Danmark. Projektet startede 1. maj 2017 og består af to arbejdspakker. De omhandler to forskellige, men direkte relaterede aspekter, der er nødvendige for fremtidig brug af miljø-DNA i dansk naturforvaltning, Arbejdspakke 1: Opbygning af en national DNA-referencedatabase, Arbejdspakke 2: Udvikling af miljø-DNA felt-, laboratorie- og analysemetodik til at kortlægge biodiversitet i terrestriske økosystemer. Denne årsrapport dækker den første halvdel af DNAmark-projektet.
I Arbejdspakke 1 udvikler vi arbejdsgange til at generere referencedata for så forskellige prøvetyper som muligt og genererer referencedata for mindst 1000 danske arter på tværs af taksonomiske grupper og af særlig interesse for dansk naturovervågning. Til at hjælpe os med at udvælge og indsamle de første 1000 arter, har vi dannet en komité af interesserede parter, som består af blandt andet kuratorer, miljø-DNA-forskere og naturforvaltere. Komitéen er vokset til 48 medlemmer, hvoraf mange har deltaget på vores årlige komitémøder. Da vi har fokus på at dække så mange taksonomiske ordner som muligt, har indsamlingen af prøver været tidskrævende. Vi har indsamlet prøver fra ca. 670 danske arter fordelt på 126 forskellige taksonomiske ordner, som blandt andet dækker ferskvandsindikatorarter, sjældne orkideer og ældre museumseksemplarer. De resterende arter er planlagt indsamlet ultimo 2018. I laboratoriet er nye protokoller udviklet, således at vi kan generere DNA-referencedata fra forskellige taksonomiske grupper og prøvetyper. Af de omkring 670 indsamlede prøver er 545 under bearbejdning eller færdigbearbejdede i laboratoriet, og referencedata for de første arter er gjort tilgængeligt via NCBI Genbank. Vi er i gang med at udvikle en platform hvorfra data kan tilgås, som skaber overblik og adgang til det generede referencedata i NCBI Genbank. Der er stor national og international interesse for DNAmark DNA-referencedatabasen. Vi har haft besøg af ledende personer fra Natural History Museum i London, Translational Genomics of Biodiversity Center (Tyskland), Naturalis Biodiversity Center (Holland) og Smithsonian Institute’s Museum of Natural History (USA) samt etableret samarbejder med blandt andre KEW Gardens (London), DTU Aqua (Danmarks Tekniske Universitet) og Institut for Geovidenskab og Naturforvaltning (Københavns Universitet).
I Arbejdspakke 2 går arbejdet med at udvikle miljø-DNA til et praktisk værktøj til objektivt at opgøre biodiversitet fremad. Målet er at man i fremtiden kan anvende dette værktøj i prioritering af arealer og i evaluering af virkemidler i naturbeskyttelse. I denne arbejdspakke er ansat to medarbejdere, en postdoc og en videnskabelig assistent. I projektets første år fokuserer vi på 10 udvalgte prøveflader ud af BioWide-projektets 130 prøveflader. De ti udvalgte prøveflader repræsenterer tilsammen forskellige økosystemer fra simple til komplekse samt forskellige abiotiske miljøer. Det er i disse ti udvalgte prøveflader, at vi indsamler prøver fra forskellige substrater (levesteder), som vi har grund til at tro har forskellige arter tilknyttet. Fx har jord forskellige plantearters dele, døde plantedele som bladførne og dødt ved i forskellige størrelsesklasser, samt gødning fra store dyr. Fra de 10 fokusprøveflader har vi taget prøver fra mere end 300 forskellige substrater. Det giver et gennemsnit på mere end 30 substrater per prøveflade. Dette gennemsnit dækker over meget stor variation fra simple til komplekse økosystemer. Fra disse prøver er DNA ekstraheret og sekvenseret. Vi har modtaget sekvensdata for DNA-markører målrettet svampe, insekter og generelle eukaryoter (ikke bakterier). Data for planter og leddyr er på vej fra Sekvenseringscenteret på Statens Naturhistoriske Museum (SNM). Vi er langt med at udvikle et analyseformat på basis af de første data, som vi derefter kan anvende på de øvrige DNA-markører. Vi vil således snart være i stand til at besvare spørgsmål om hvilke substrater det er mest omkostningseffektivt af inkludere i en praktisk indsamlingsprotokol.
Vores målsætning er, at DNAmark-projektet skal være synligt for og anvendes af omverden, både internt af komitéen af interesserede parter, men også for andre nationale og internationale fagpersoner og dele af offentligheden, som kunne have interesse i og gavn af projektet. Vi formidler derfor projektet via vores hjemmeside, via Instagram, til videnskabelige konferencer, i videnskabelige publikationer samt populærvidenskabeligt ved præsentationer og i nyhedsbreve.
Status 2017
Projektet startede 1. maj 2017 og består af to overordnede arbejdspakker, Arbejdspakke 1 og Arbejdspakke 2, der omhandler to forskellige, men direkte relaterede aspekter, der er nødvendige for fremtidig brug af miljø-DNA i dansk naturforvaltning: (i) Opbygning af en dansk DNA-referencedatabase og (ii) udvikling af miljø-DNA felt-, laboratorie- og analysemetodik til at kortlægge multi-taxon biodiversitet (inklusiv artsrige, men vanskelige grupper som spindlere, insekter og svampe) i terrestriske økosystemer langs hele gradienten i successionstrin fra klitlavning over højmose til gamle krat og urskov. Et kort resumé af hver arbejdspakkes fremskridt og resultater for de første seks måneder af projektperioden følger nedenfor.
I Arbejdspakke 1 har vi fokus på at udvikle protokoller og arbejdsgange til at generere referencedata for så forskellige prøvetyper som muligt og på at generere referencedata for mindst 1000 udvalgte danske arter på tværs af taksonomiske grupper og af særlig interesse for dansk naturovervågning.
DNAmark DNA-referencedatabasen er kommet godt fra start. Vi har ansat tre medarbejdere: En videnskabelig assistent, en bioinformatiker og en projektkoordinator. Vi har i samarbejde med vores ‘komité af interesserede parter’ bestående af blandt andet kuratorer, miljø-DNA-forskere og naturforvaltere udvalgt de første ca. 800 danske arter til DNA-referencedatabasen. Heraf har vi indsamlet prøver fra ca. 230 arter. I laboratoriet er nye protokoller ved at blive udviklet, således at vi kan generere DNA-referencedata fra forskellige taksonomiske grupper og prøvetyper. DNA er blevet ekstraheret fra 137 prøver, herunder 33 pattedyr, 39 fugle, 42 fisk, 10 insekter, 8 padder og 5 krybdyr, og nogle af disse DNA-ekstrakter er klargjort til sekventering. Endvidere har vi fået sekvensdata fra andre forskningsprojekter, for eksempel fra ‘The Bird 10,000 Genomes (B10K)’-projektet, hvorfra vi har genereret referencedata for 26 fuglearter.
En ph.d.-studerende finansieret gennem det Europæiske Marie Curie-netværk Plant.ID (http://www.plantid.uio.no/) bliver ansat fra april 2018. Den studerende skal udforske anvendt brug af denne nye type DNA-referencedatabase til at detektere plantearter i miljø-DNA-studier. Der er stor international interesse for DNAmark DNA-referencedatabasen, og vi har haft besøg af ledende personer fra for eksempel Natural History Museum i London, Translational Genomics of Biodiversity Center (Tyskland), Naturalis Biodiversity Center (Holland) og Smithsonian Institute’s Museum of Natural History (USA).
I Arbejdspakke 2 skrider arbejdet med at udvikle miljø-DNA til et praktisk værktøj til objektivt at opgøre biodiversitet og dermed bruge dette værktøj i prioritering og evaluering af virkemidler i naturbeskyttelsen også støt fremad. I denne arbejdspakke er ansat to nye medarbejdere, en postdoc og en videnskabelig assistent. Ved projektets start er 10 af BioWide-projektets 130 prøveflader blevet udvalgt. De ti udvalgte prøveflader repræsenterer meget forskellige økosystemer fra simple til komplekse og med meget forskellige abiotiske miljøer. Det er i disse ti udvalgte prøveflader, at vi er gået i dybden med at indsamle prøver fra forskellige substrater. I august måned startede feltarbejdet, som består af intensiv indsamling af de første prøver til DNA-ekstraktion. Der blev indsamlet prøver fra forskellige substrattyper såsom jord, levende træers bark, ved og blade, bladførne, grenførne, dødt ved samt urteagtige planters rhizomer, blade og blomster. Oparbejdning af prøverne er afsluttet og ekstraktion af DNA i fuld gang. De organismegrupper der vil være fokus på er især svampe, insekter, nematoder, protozoer og bakterier.
Vi ønsker, at DNAmark-projektet skal være synligt for omverden, både internt på Statens Naturhistoriske Museum, for komitéen af interesserede parter, men også for andre fagpersoner og dele af offentligheden, som kunne have interesse i og gavn af projektet. Vi har derfor lavet en hjemmeside på dansk og engelsk, fået et logo samt oprettet en Instagram-profil for at kunne dele projektets fremskridt ved hjælp af nyheder og billeder. Desuden har vi formidlet projektet på videnskabelige konferencer, i videnskabelige publikationer samt populærvidenskabeligt ved præsentationer, i radio og på film.
Artikler fra projektet
Projekt info
Projektdeltagere
Tom Gilbert og Kristine Bohmann, EvoGenomics, Statens Naturhistoriske Museum, Københavns Universitet
Hans Henrik Bruun, Biologisk Institut, Københavns Universitet
Rasmus Ejrnæs, Bioscience, Aarhus Universitet
Anders J. Hansen, Center for GeoGenetik, Statens Naturhistoriske Museum, Københavns Universitet
Rasmus Kjøller og Tobias G. Frøslev, Biologisk Institut, Københavns Universitet
Kontaktperson
Thomas Gilbert, Statens Naturhistoriske Museum, Københavns Universitet
Projektforløb
2017 – 2020
Hjemmeside